Ya se encuentra on line y activo el sitio web del Proyecto COVID-FEDERAL, CORR01 en donde se muestran resultados de un monitoreo a tiempo real y predicción del avance de la pandemia COVID-19 en Argentina utilizando un sistema de asimilación de datos combinado con técnicas de aprendizaje automatizado basado en ensambles.
El sistema desarrollado y el sitio web que muestra los resultados son producto del proyecto «Sistema de monitoreo y predicción del COVID-19 en la provincia de Corrientes usando asimilación de datos», cuyo financiamiento es otorgado por la Agencia Nacional de Promoción Científica Tecnológica (ANPCyT).
El sitio web es de acceso y consulta libre en esta dirección:
http://covid19.unne.edu.ar
Sus realizadores integran un equipo interdisciplinario de la Universidad Nacional del Nordeste y el Conicet compuesto por físicos, matemáticos y bioquímicos, liderados por el doctor Manuel Pulido.
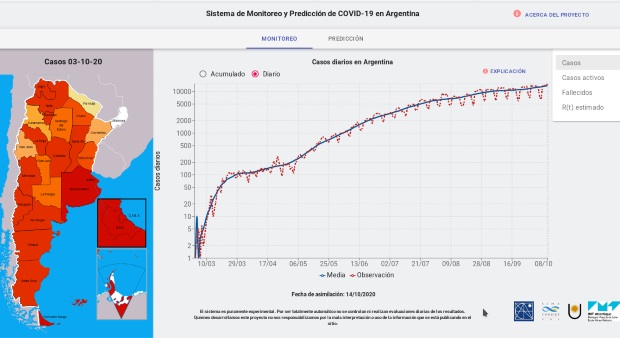
El sistema permite el aprendizaje de parámetros de la epidemia a través de los datos que se procesan. Una de las informaciones aportada por el trabajo, es la estimación del R efectivo ó tasa de contagio del coronavirus a nivel nacional y provincial. Antes del inicio de la cuarentena en el mes de marzo, la tasa era 3,5 es decir un infectado contagiaba a 3,5 personas. La implementación de la primera etapa de la cuarentena logró reducir considerablemente esa tasa a 0,5. En la actualidad, el R efectivo está levemente por arriba de 1.
La importancia de tener la estimación diaria de esta tasa es clave, ya que cuando se alcance un valor por debajo de 1, la epidemia se controla y el número de casos disminuirá con el tiempo. Por el contrario, si la tasa de contagio está por encima de 1, significa que la cantidad de infectados continuará creciendo.
¿Qué datos se utilizan para el monitoreo?, ¿A qué hora del día se cargan?
El sistema trabaja a tiempo real bajando los datos diarios y procesándolos en forma automática. Los datos se bajan y procesan durante la noche, para asegurar su actualización. Se utiliza la base de datos del Ministerio de Salud de la Nación y scraping del informe diario.
¿Qué es un Sistema de Asimilación de Datos?. El sistema está basado en una técnica físico-estadística que permite combinar información de datos existentes de distintas fuentes con información de un modelo epidemiológico.
Esta metodología permite determinar de manera objetiva diferentes parámetros relacionados con la propagación de la enfermedad, así como también determinar la precisión con la que los mismos pueden ser conocidos.
De esta manera se puede obtener información acerca de cómo se comporta la enfermedad, y cómo impactan diferentes medidas de distanciamiento social sobre la propagación de la misma.
Por otra parte la información obtenida permite realizar proyecciones de la evolución futura de la epidemia junto con una cuantificación del nivel de incertidumbre asociada a las mismas. En el proceso de estimación se considera las posibles fuentes de incertidumbre, tanto las asociadas a las fuentes de información como también aquellas provenientes de las predicciones del modelo epidemiológico. Para considerar las incertezas en la estimación se trabaja con una técnica de Monte Carlo que utiliza un conjunto numeroso de posibles escenarios de propagación de la enfermedad.
Modelo epidemiológico. El modelo epidemiológico utilizado en el sistema de asimilación de datos es del tipo SEIRHD (suceptibles – expuestos – infectados – recuperados – hospitalizados y fallecidos por sus sigla en inglés). En donde la población se divide en fracciones o compartimentos. Dichas fracciones corresponden a los susceptibles (aquellos que no han estado en contacto con el virus), los expuestos (aquellos que han tenido contacto con una persona infectada pero aún no cursan la enfermedad), los infectados (aquellos que se encuentran cursando la enfermedad con diferentes sintomatologías), recuperados (aquellos que habiendo estado infectados ya se encuentran recuperados y por tanto se supone inmunes a la enfermedad), hospitalizados (aquellos que por la severidad de sus requisitos requieren algún tipo de internación hospitalaria) y los fallecidos (aquellos que murieron como consecuencia directa de la enfermedad).
El modelo SEIRHD permite estimar la evolución en el tiempo de la cantidad de personas que se encuentra en cada categoría a través de un sistema de ecuaciones diferenciales acopladas.
Este sistema de ecuaciones incluye parámetros que controlan aspectos tales como la tasa de contagios o la mortalidad del virus. Estos parámetros dependen de factores externos como el nivel de distanciamiento social o las propiedades del virus y su valor no es conocido con precisión y además puede variar de acuerdo a la localidad o a las medidas de distanciamiento social.
La información obtenida a través de los datos, es utilizada para el aprendizaje de los parámetros del modelo, como también para la estimación de las variables del sistema.
Equipo de Investigadores. El grupo de trabajo que participo de este proyecto está integrado por los investigadores: Manuel Pulido (Director, UNNE-CONICET), Juan Ruiz (Investigador, UBA-CONICET); Magdalena Lucini (UNNE-CONICET), Santiago Rosa (Pasante graduado. Lic FaMAF, UNCordoba); Fido García (CPA, CIMA-CONICET), Agustina Quiros (Pasante. Est. Lic. En Sistemas, FaCENA, UNNE), Angel Romero y Gaston Contardi (Pasantes. Est. Lic. En Fisica FaCENA, UNNE), Tadeo Cocucci (Becario CONICET. FaMAF UNCordoba, FaCENA UNNE), Santiago Wiedman (Est. Lic. En Fisica, FaCENA, UNNE), Gerardo Andino (Laboratorio Central Prov. Corrientes), Viviana Gutnisky (Laboratorio Central Prov. Corrientes), Natalia Ruiz Dias (Laboratorio Central Prov. Corrientes).
Se conformó una red de colaboración con distintos grupos que trabajan en asimilación de datos para COVID-19, entre los que se destacan un grupo de investigadores de Francia coordinado por Pierre Tandeo del IMT Atlantique; Juan Aparicio (UNSa, CONICET) y Geir Evensen (NORCE, Noruega)

















